Quel est ce projet en quelques mots ?
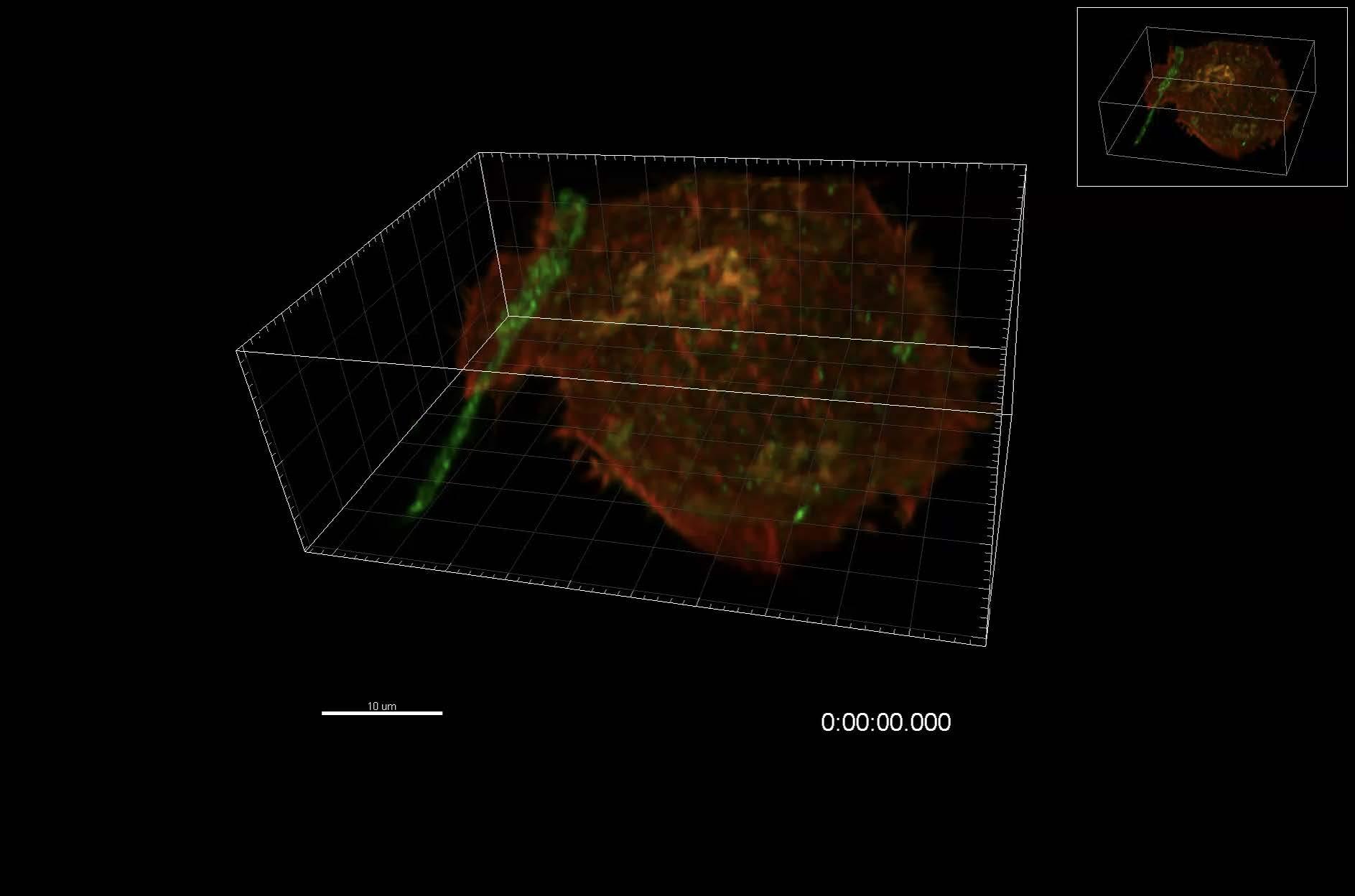
« Naviscope a pour objectif de développer des méthodologies permettant de manipuler des images complexes observées en microscopie, aux échelles intracellulaire (à l’intérieur d’une cellule) et multicellulaire (concernant un groupe de cellules). » explique Charles Kervrann, coordinateur du Défi Naviscope. « L’écrasante quantité de données à analyser fait que, bien souvent, on est submergé, à des échelles spatiales qui rendent difficile l’interprétation. »
Il s’agit de séries temporelles de volumes 3D. Ce qu’on appelle aussi imagerie 4D (avec le temps en quatrième dimension).
Le projet a pour but de concevoir un logiciel permettant d’interagir et de visualiser des données complexes 3D + temps, en étant guidé par l’apprentissage via des annotations stockées dans des bases de données. Aucun logiciel ne peut faire cela dans son entièreté.
Comment est né votre projet ?
« Quand on discute avec les autres partenaires qui acquièrent des images en microscopie 4D, une grande difficulté rencontrée est de pouvoir naviguer et explorer toutes ces données. »
La philosophie des Défis Inria est d’essayer de construire un dessein commun qui ne pourrait pas être abordé par une équipe isolée. La grande force de ces projets est de mettre en partage les savoir-faire de différentes équipes-projets, en l’occurrence, pour Naviscope, en « computer graphics, visualization, machine learning, optics, cell biology ».
« Nous développons des méthodes de traitement d’image 3D mais la visualisation n’est pas le cœur de métier de notre équipe (SERPICO), d’où la collaboration avec HYBRID et AVIZ en particulier afin qu’ils apportent des compétences spécifiques pour répondre à nos besoins. De leur côté, ces équipes y trouvent aussi leur compte car elles vont développer des approches méthodologiques originales en lien avec de nouvelles thématiques applicatives et donc de nouveaux horizons. »
En quoi votre projet est-il novateur ?
Naviscope vise également à incorporer les principes de l’apprentissage, relativement absents dans les approches conventionnelles de visualisation de données. Une des originalités du projet est d’essayer d’une manière ou d’une autre de concevoir un outil destiné à la fois à guider l’exploration des données avec des techniques d’apprentissage et à permettre à un utilisateur d’annoter des données.
Quels sont les premiers résultats ?
« Le projet Naviscope a commencé il y a deux ans et les résultats que l’on peut d’ores et déjà présenter sont donc issus d’une année de réflexion intense et conceptuelle suivie d’une année plus spécifiquement de mise en pratique. »
Il est déjà possible de permettre aux biologistes de s’immerger avec un casque et de manipuler des données grâce à l’intégration de données dérivées (numériques, symboliques), ce qui constitue une première partie probante des résultats attendus. Naviscope commence également à produire des résultats sur les questions d’annotation de données pour constituer des bases d’apprentissage.
« Dans deux ans, une grande partie des objectifs sera opérationnelle et je pense que nous serons arrivés au stade de la preuve de concept, c’est-à-dire au stade de l’évaluation par des bêta-testeurs (biologistes) qui pourront certifier ou non que les dispositifs fonctionnent. »
Quels sont les applications ou les usages de vos découvertes ? Y a-t-il des domaines particuliers pour lesquels vous travaillez ?
Notre objectif final est que les biologistes puissent faire des découvertes en biologie grâce à un outil comme le nôtre.
« Une autre retombée potentielle serait d’élaborer des outils beaucoup plus simples pour enseigner aux étudiants en biologie. Voir des molécules en interaction, comprendre la dynamique des virus dans la cellule avec un casque d’immersion par exemple. »
Quelles sont les prochaines étapes ?
« Nous allons regrouper toutes ces briques logicielles dans un cadre unifié d’interopérabilité pour que la manipulation des données soit simple et interactive, avec une documentation en ligne. »
Coordinateur : C. Kervrann.
Partenaires : équipe Inria AVIZ (Saclay) ; équipe Inria BEAGLE (Lyon), équipe Inria HYBRID (Rennes), équipe Inria MORPHEME (Sophia-Antipolis) ; équipe Inria MOSAIC (Lyon), équipe Inria PARIETAL (Saclay), équipe Inria SERPICO (Rennes) ; Unité MaIAGE INRAE (Jouy-en-Josas) ; Institut Curie Paris (équipes STED et LOCCO) ; Institut Pasteur Paris (équipe DBC) (Paris).