COVID19 Disease Map : outil interdisciplinaire, interactif et open source
Près de deux ans de travail, un consortium de 277 scientifiques de 130 institutions dans 30 pays aura permis de créer une plate-forme interactive – une carte virtuelle – compilant l’ensemble des données disponibles sur les interactions moléculaires entre le virus de la Covid-19 et notre organisme, et leur rôle dans les mécanismes complexes de la maladie : processus d’infection, voies biologiques et types de cellules affectés, prolifération virale, réponse inflammatoire…
COVID-19 Disease Map, a computational knowledge repository of virus-host interaction mechanisms
L’outil baptisé COVID19 Disease Map a ainsi bénéficié d’une collaboration scientifique exceptionnelle couvrant tous les champs d’expertise nécessaires à la compréhension du virus :
- les aspects cliniques et moléculaires de la maladie ;
- la caractérisation des interactions moléculaires et la construction des diagrammes de biologie des systèmes représentant les mécanismes de la maladie ;
- l’exploration de la littérature médicale ;
- l’intégration de diverses bases de données et les compétences bio-informatiques ;
- la modélisation informatique pour analyser les perturbations liées aux maladies à l’aide d’approches de biologie des systèmes.
Ce défi ne s’arrête pas là : cette carte virtuelle accessible en open source s’enrichit au fur et à mesure de l’avancée des connaissances sur la Covid-19 afin d’améliorer en temps réel notre compréhension de la maladie et d'accélérer le développement de diagnostics et de thérapies adaptées.
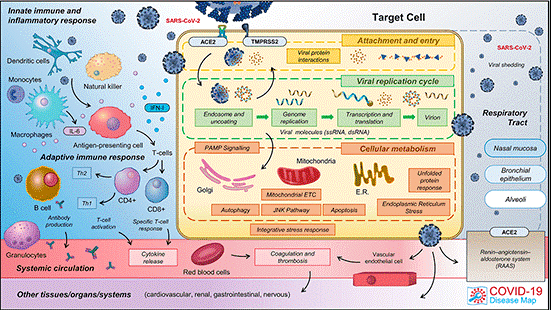
COVID19 Disease Map : Carte de l’ensemble des mécanismes moléculaires impliqués dans la maladie
CaSQ, pour une analyse dynamique
Au-delà de l’analyse statique des voies biologiques impliquées, l’outil permet également une visualisation interactive : l’utilisateur, en agissant sur divers éléments, protéines, gènes… observe en direct l’effet produit par l’activation ou la suppression en question.
Il permet ainsi aux scientifiques de simuler et d’analyser l’effet de perturbations – une réaction inflammatoire, la prolifération du virus ou l’effet d’un médicament – grâce à des modèles informatiques interopérables. Ce point représente d’ailleurs un enjeu majeur quant à l’exploitation efficiente de l’outil. Car rendre compatibles entre eux des dizaines de logiciels et de plates-formes développés à travers le monde est un challenge bien plus ardu qu’il n’y paraît.
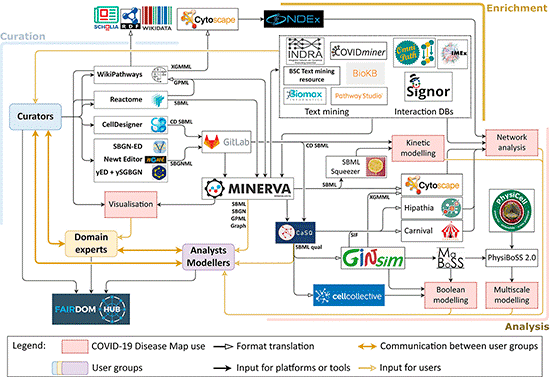
Ecosystème de plateformes informatiques et logiciels constituant la COVID19 Disease Map
Parmi les logiciels exploités et intégrés se trouve CaSQ, un outil développé par Sylvain Soliman, chercheur de l’équipe Lifeware du centre Inria de Saclay et Anna Niarakis. CaSQ traduit les cartes d’interactions moléculaires avec des règles mathématiques afin d’établir un modèle dynamique dans un format standard (SBML-qual) compatible avec plusieurs outils de simulations in silico et exécutables à grande échelle. Ainsi en une poignée de secondes, CaSQ produit un modèle dynamique à partir de données statiques.
Ces modèles dynamiques sont ensuite analysés par des logiciels spécialisés, en particulier GINsim, développé par Aurélien Naldi, chercheur de l’équipe LIfeware. GINsim interprète les règles mathématiques générées par CaSQ et permet de prédire le comportement du modèle au cours du temps. Cette analyse dynamique repose en grande partie sur des méthodes formelles en constante évolution, souvent implémentées dans des logiciels spécifiques. Pour faciliter les analyses combinant plusieurs logiciels, Aurélien Naldi et d’autres chercheurs ont mis en place le Notebook CoLoMoTo : une interface unifiée pour une quinzaine de logiciels et mettant l’accent sur la reproductibilité des analyses.
Au sein de l’équipe Lifeware, à chacun son expertise :
- Anna Niarakis, enseignante-chercheuse en délégation, implémente les règles biologiques dans les outils et vérifie les résultats ;
- Sylvain Soliman, chercheur, programme le développement du logiciel pour son exploitation au sein de l’outil COVID19 Disease Map ;
- Aurélien Naldi, chercheur, analyse les modèles produits en utilisant les logiciels et les pipelines qu’il développe au sein de la communauté.
Prochains objectifs
Le but aujourd’hui est de permettre l’identification de différentes prédispositions à la Covid-19 ainsi que la priorisation des candidats médicaments.